Química
1 Lei dos Gases Ideais (EM13CNT101, EM13CNT204, EM13CNT205, EM13CNT207, EM13CNT303)
# Dados:
R = 8.314 # J/(mol*K), constante geral dos gases ideias
V = seq(0,22.4, length.out=50) # vetor de "Volume" (em litros), com 50 pontos
T = 298 # K, temperatura absoluta
# Equação
# pV = RT; p = RT/V
p = R*T/V
library(plotly)
library(magrittr) # biblioteca para o operador pipe "%>%"
plot_ly(x = V, y = ~R*T/V, type = 'scatter', mode='lines') %>%
layout(
title = "Transformação Isotérmica de um Gás",
xaxis = list(title = "Volume V, L", range=c(-1,24)),
yaxis = list(title = "Pressão p, Pascal")
)Sugestões:
Experimente modificar o gráfico, usando/substituindo alternativamente os comandos abaixo no trecho de código:
plot_ly(x = V, y = ~R*T/V, type = 'scatter', mode='lines', line = list(color = "red", width = 3)) %>% # Alteração da cor e largura da linha
2 Curvas de consumo de reagente e formação de produto (EM13CNT301, EM13MAT304, EM13CNT306)
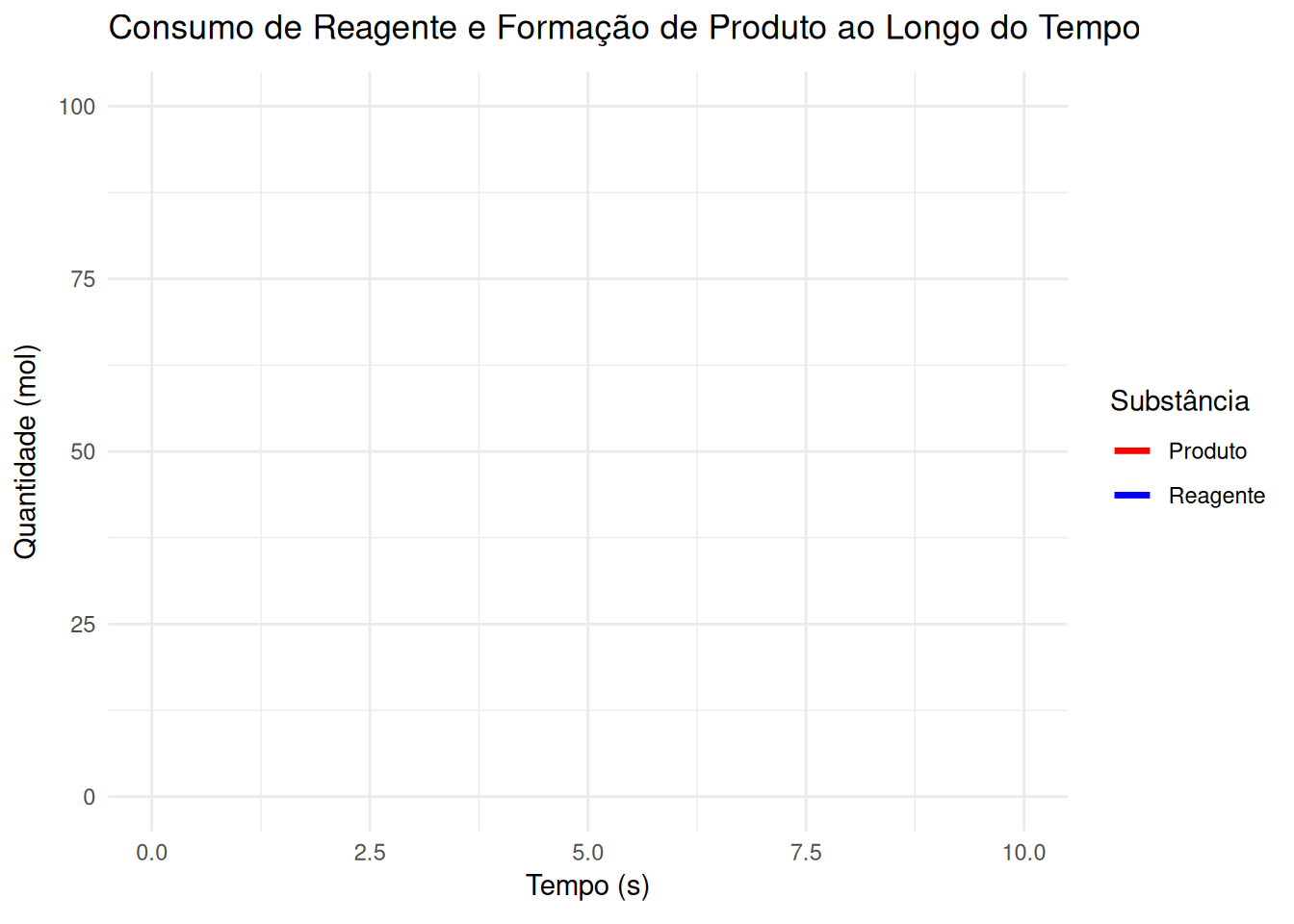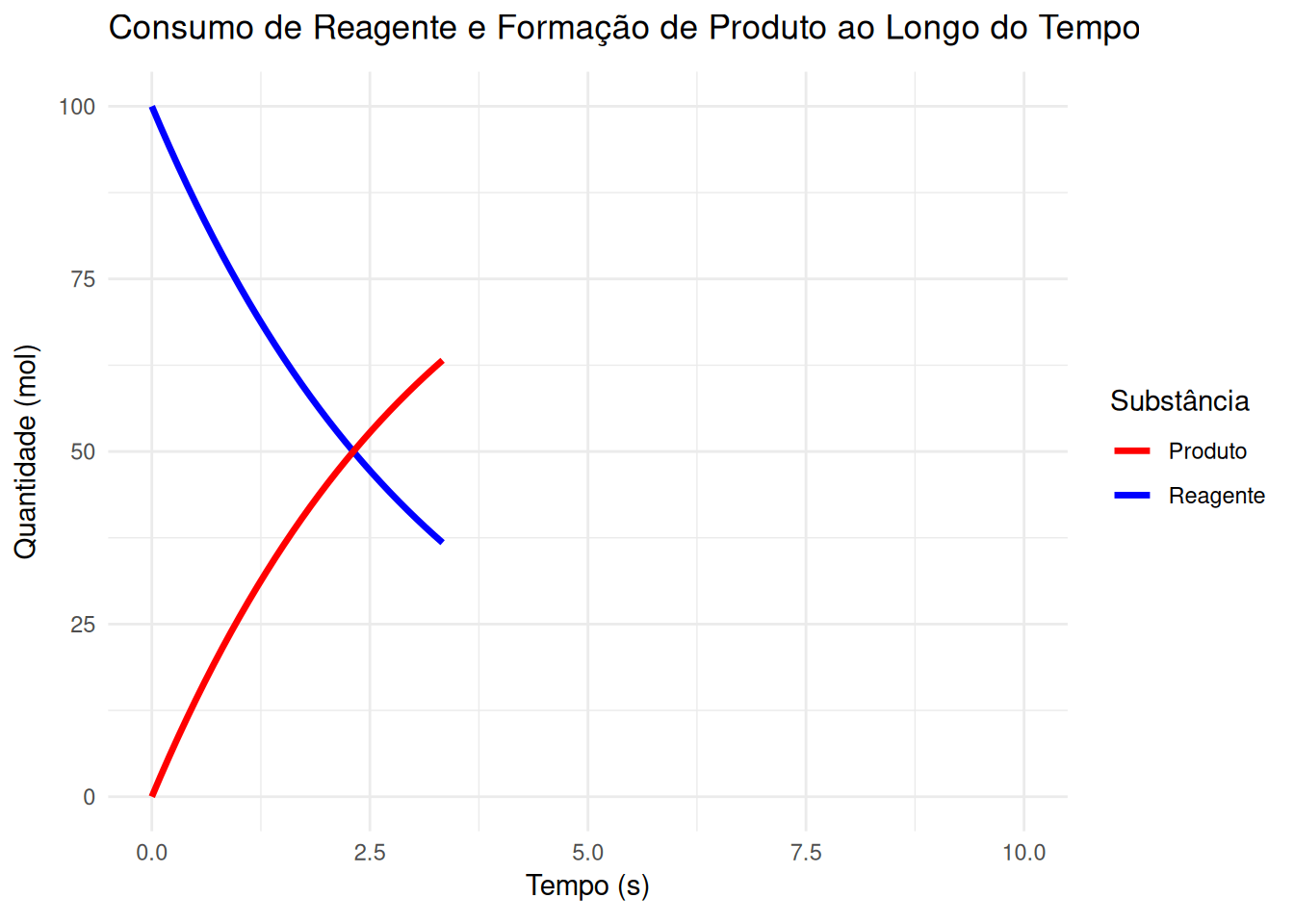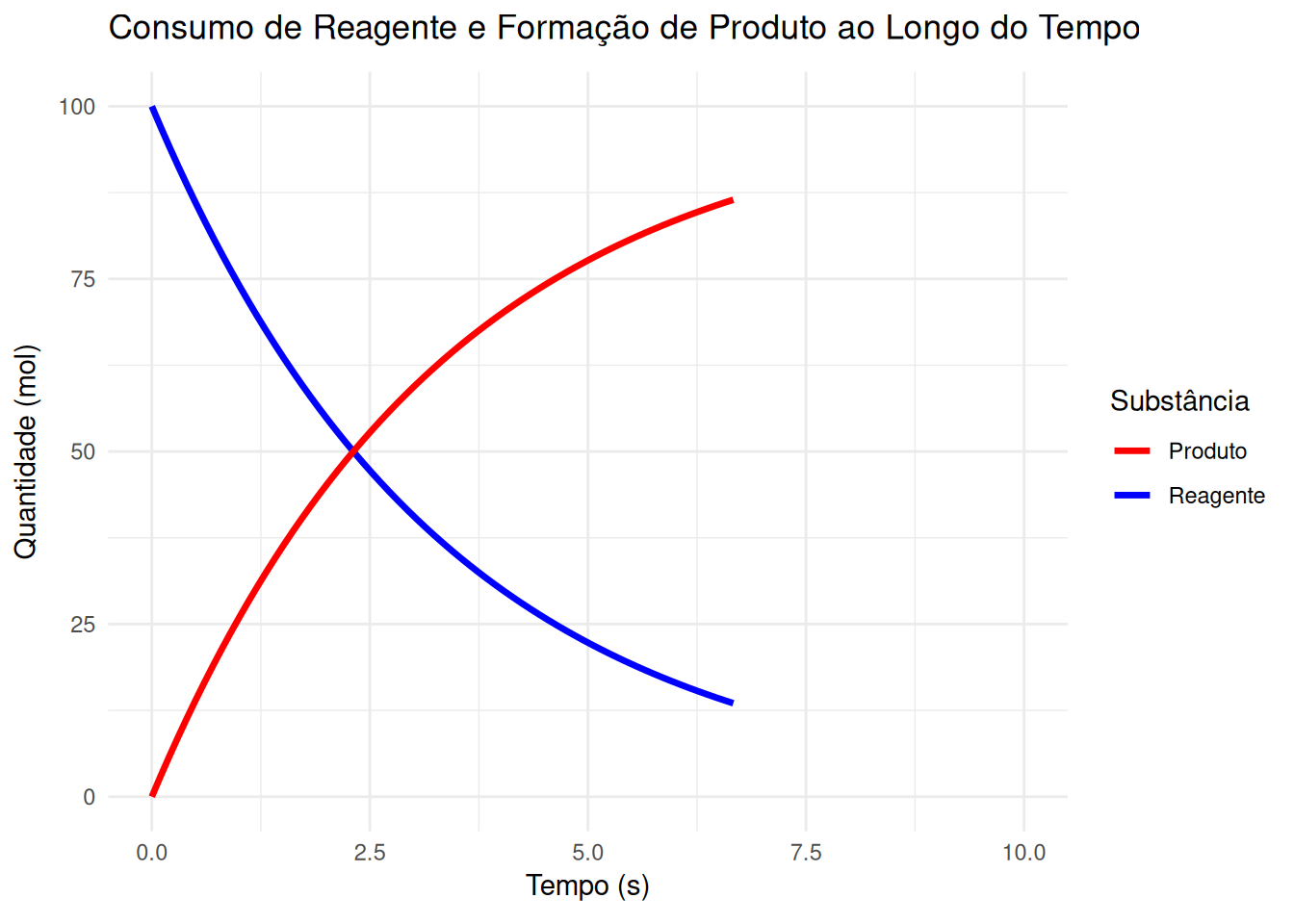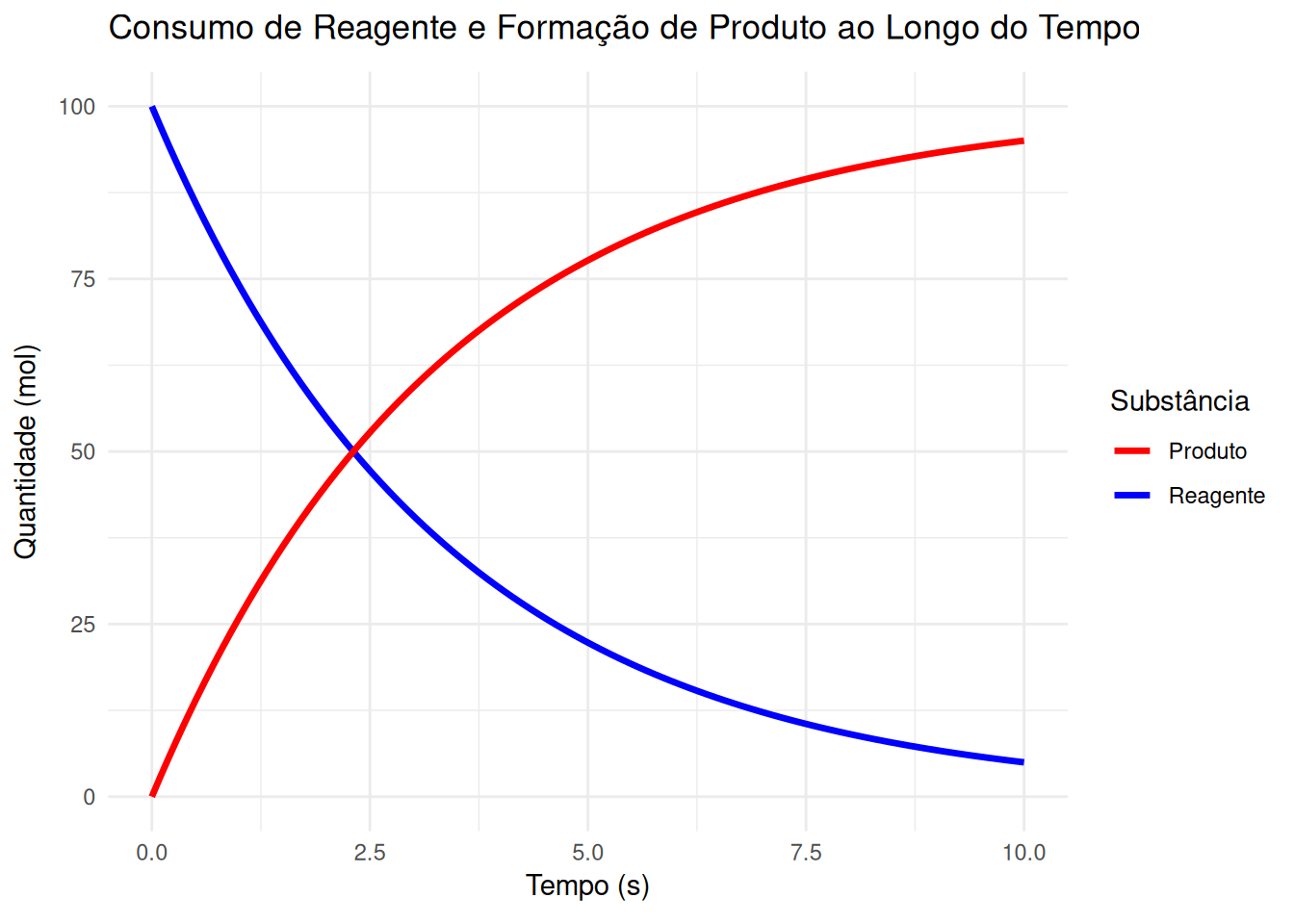
library(ggplot2)
library(gganimate)
library(gifski)
# Dados simulados de tempo, reagente e produto
tempo <- seq(0, 10, length.out = 100) # 0 a 10 segundos
reagente <- 100 * exp(-0.3 * tempo) # Decaimento exponencial
produto <- 100 * (1 - exp(-0.3 * tempo)) # Formação de produto
# Criando um dataframe para o gráfico
dados <- data.frame(Tempo = tempo, Reagente = reagente, Produto = produto)
# Gráfico animado de consumo de reagente e formação de produto
p <- ggplot(dados, aes(x = Tempo)) +
geom_line(aes(y = Reagente, color = "Reagente"), linewidth = 1.2) +
geom_line(aes(y = Produto, color = "Produto"), linewidth = 1.2) +
scale_color_manual(values = c("Reagente" = "blue", "Produto" = "red")) +
labs(title = "Consumo de Reagente e Formação de Produto ao Longo do Tempo",
x = "Tempo (s)", y = "Quantidade (mol)", color = "Substância") +
transition_reveal(Tempo) +
theme_minimal()
# Exibindo o gráfico animado
animate(p, renderer = gifski_renderer())Sugestões:
Experimente modificar o gráfico, usando/substituindo alternativamente os comandos abaixo no trecho de código:
scale_color_manual(values = c("Reagente" = "green", "Produto" = "purple")) + # Mudança nas cores
3 Equilíbrio de dissociação de ácidos e bases fracas (EM13CNT303, EM13MAT305, EM13CNT304)
library(plotly)
# Parâmetro inicial de Ka para ácido fraco e dados para pH e concentração
Ka <- 1e-5 # Constante de acidez padrão
# Valores de pH e concentrações
pH_values <- seq(0, 14, 0.1)
conc_H <- 10^(-pH_values) # Concentração H+ para cada pH
conc_OH <- 1e-14 / conc_H # Concentração OH- com base no produto iônico da água
conc_HA <- conc_H / (Ka + conc_H) # Concentração HA para ácido fraco
conc_A <- Ka * conc_HA # Concentração A-
# Data frame combinado
dados_acidez <- data.frame(pH = pH_values, conc_H = conc_H, conc_OH = conc_OH, conc_HA = conc_HA, conc_A = conc_A)
# Gráfico combinado interativo
plot_ly(dados_acidez) %>%
add_lines(x = ~pH, y = ~conc_H, name = "Concentração H+", line = list(color = "blue")) %>%
add_lines(x = ~pH, y = ~conc_OH, name = "Concentração OH-", line = list(color = "red")) %>%
add_lines(x = ~pH, y = ~conc_HA, name = "Concentração HA (ácido)", line = list(color = "purple")) %>%
add_lines(x = ~pH, y = ~conc_A, name = "Concentração A- (base conjugada)", line = list(color = "green")) %>%
layout(
title = "Curvas de Acidez, Basicidade e Dissociação de Ácido",
xaxis = list(title = "pH"),
yaxis = list(title = "Concentração (mol/L)", type = "log"),
sliders = list(
list(
active = 5,
currentvalue = list(prefix = "Ka = "),
steps = lapply(seq(1e-7, 1e-3, length.out = 10), function(k) {
list(label = format(k, scientific = TRUE), method = "update",
args = list(list(
y = list(
10^(-pH_values), # Atualizar H+
1e-14 / 10^(-pH_values), # Atualizar OH-
10^(-pH_values) / (k + 10^(-pH_values)), # Atualizar HA
k * (10^(-pH_values) / (k + 10^(-pH_values))) # Atualizar A-
)
)))
})
)
)
)Sugestões:
Experimente modificar o gráfico, usando/substituindo alternativamente os comandos abaixo no trecho de código:
add_lines(x = ~pH, y = ~conc_H, name = "Concentração H+", line = list(color = "blue", width = 2)) %>% # Aumento da espessura da linha
add_lines(x = ~pH, y = ~conc_OH, name = "Concentração OH-", line = list(color = "red", width = 2)) %>% # Aumento da espessura da linha
add_lines(x = ~pH, y = ~conc_HA, name = "Concentração HA (ácido)", line = list(color = "purple", width = 2)) %>% # Aumento da espessura da linha
add_lines(x = ~pH, y = ~conc_A, name = "Concentração A- (base conjugada)", line = list(color = "green", width = 2)) %>% # Aumento da espessura da linha
4 Decaimento radioativo e meia-vida (EM13CNT306, EM13MAT305, EM13CNT301)
library(plotly)
# Parâmetros iniciais
N0 <- 100 # Quantidade inicial
tempo <- seq(0, 50, by = 0.5) # Intervalo de tempo
# Função de decaimento
decaimento_radioativo <- function(t, meia_vida) {
N0 * 0.5^(t / meia_vida)
}
# Gráfico com plotly
fig <- plot_ly(x = ~tempo, y = ~decaimento_radioativo(tempo, 10), type = 'scatter', mode = 'lines', name = "Decaimento")
# Adicionando o slider
fig <- fig %>% layout(
title = "Decaimento Radioativo",
xaxis = list(title = "Tempo"),
yaxis = list(title = "Quantidade Restante"),
sliders = list(list(
currentvalue = list(prefix = "Meia-vida = "),
steps = lapply(seq(5, 30, by = 5), function(meia_vida) {
list(label = as.character(meia_vida), method = "update",
args = list(list(y = list(decaimento_radioativo(tempo, meia_vida)))))
})
))
)
figSugestões:
Experimente modificar o gráfico, usando/substituindo alternativamente os comandos abaixo no trecho de código:
args = list(list(y = list(decaimento_radioativo(tempo, meia_vida))), list(line = list(color = 'green'))))
5 Decomposição de peróxido de hidrogênio (EM13CNT301, EM13MAT305, EM13CNT303)
library(plotly)
# Parâmetros iniciais
H2O2_0 <- 1 # Concentração inicial de H2O2
tempo <- seq(0, 10, 0.1) # Intervalo de tempo
# Função de decomposição
decomposicao <- function(t, k) H2O2_0 * exp(-k * t)
# Gráfico com slider
fig <- plot_ly(x = ~tempo, y = ~decomposicao(tempo, 0.3), type = 'scatter', mode = 'lines') %>%
layout(
title = "Decomposição de H2O2",
xaxis = list(title = "Tempo (horas)"),
yaxis = list(title = "Concentração de H2O2 (mol/L)"),
sliders = list(list(
currentvalue = list(prefix = "k = "),
steps = lapply(seq(0.1, 1, 0.1), function(k) {
list(label = k, method = "update", args = list(list(y = list(decomposicao(tempo, k)))))
})
))
)
figSugestões:
Experimente modificar o gráfico, usando/substituindo alternativamente os comandos abaixo no trecho de código:
list(label = k, method = "update", args = list(list(y = list(decomposicao(tempo, k))), list(line = list(color = 'red'))))
6 Lei de Proust e estequiometria de reação em representação 3D (EM13CNT101, EM13CNT202, EM13CNT201)
library(plotly)
# Quantidades iniciais de reagentes
H2_mol <- 0:5 # Quantidade de H2 em mol
O2_mol <- 0:5 # Quantidade de O2 em mol
# Função para calcular H2O formado com base na estequiometria 2:1:2
formar_H2O <- function(H2, O2) {
min(2 * H2, O2) / 2
}
# Dados para o gráfico
dados <- expand.grid(H2 = H2_mol, O2 = O2_mol)
dados$H2O <- mapply(formar_H2O, dados$H2, dados$O2)
# Gráfico interativo com sliders para H2 e O2
fig <- plot_ly(dados, x = ~H2, y = ~O2, z = ~H2O, type = 'mesh3d', mode = 'markers') %>%
layout(
title = "Estequiometria de Reação (Lei de Proust)",
scene = list(
xaxis = list(title = "Quantidade de H2 (mol)"),
yaxis = list(title = "Quantidade de O2 (mol)"),
zaxis = list(title = "Quantidade de H2O formado (mol)")
)
)
figSugestões:
Experimente modificar o gráfico, usando/substituindo alternativamente os comandos abaixo no trecho de código:
fig <- plot_ly(dados, x = ~H2, y = ~O2, z = ~H2O, type = 'mesh3d', mode = 'markers',
marker = list(color = ~H2O, colorscale = 'Viridis', size = 10)) %>%
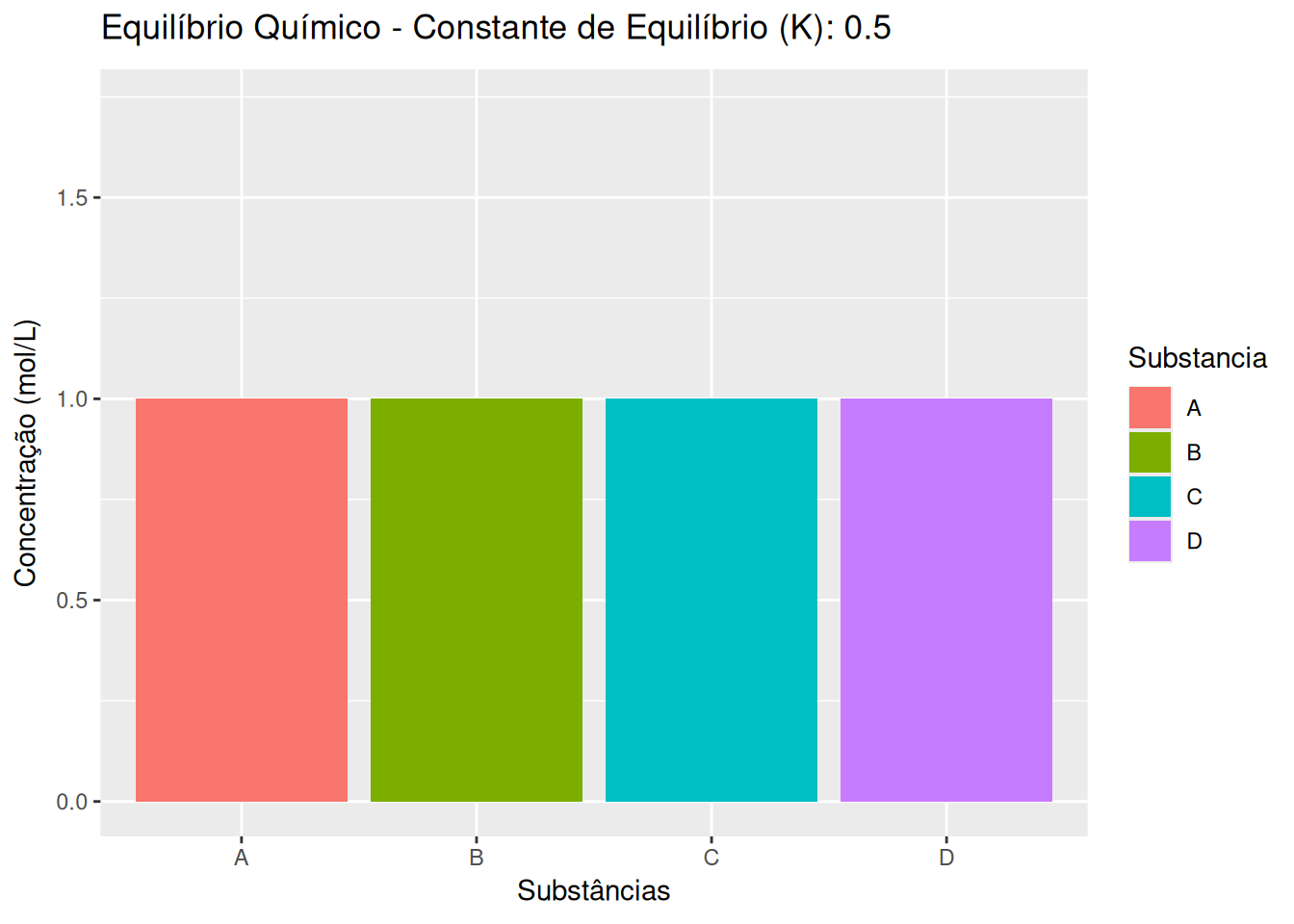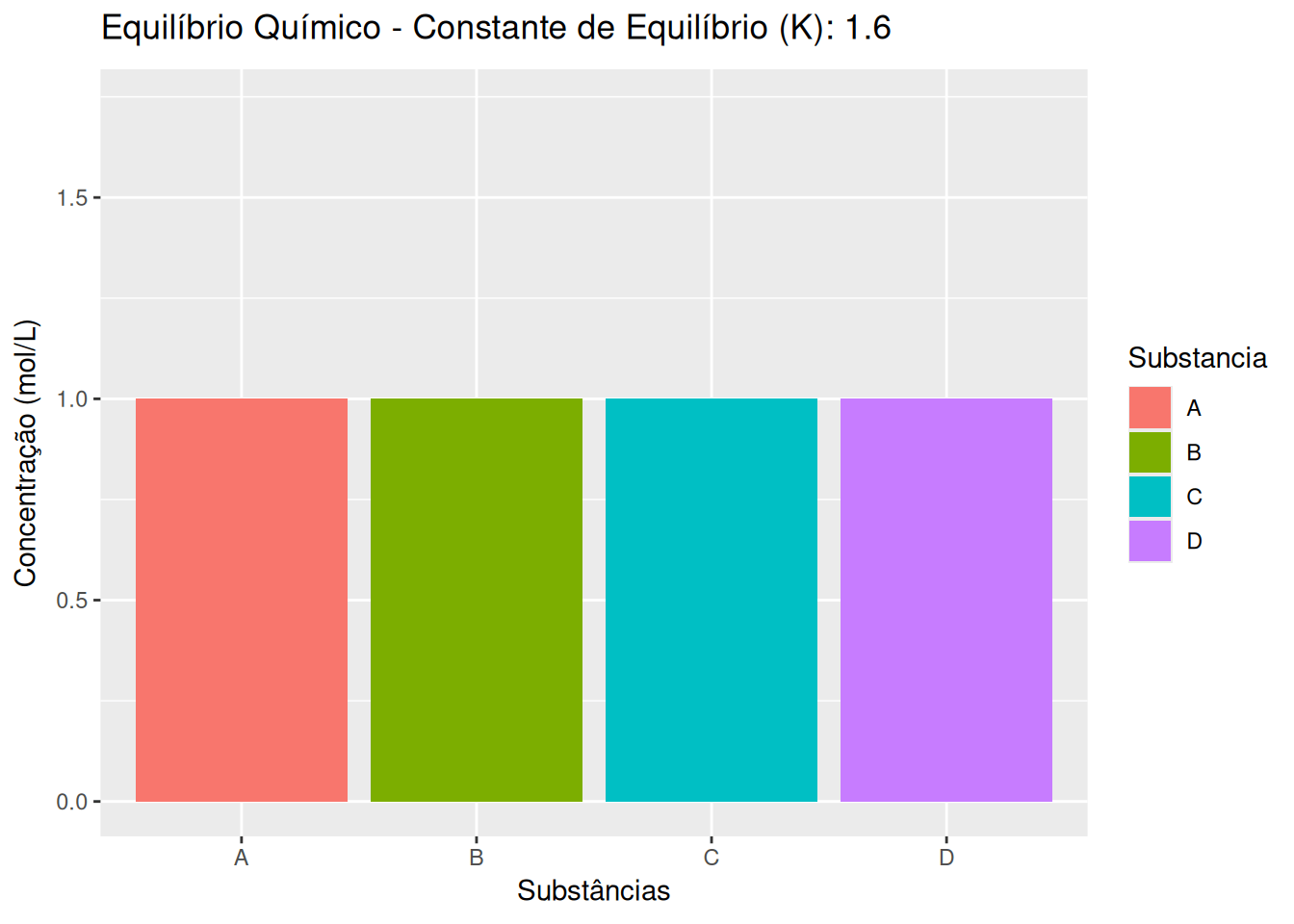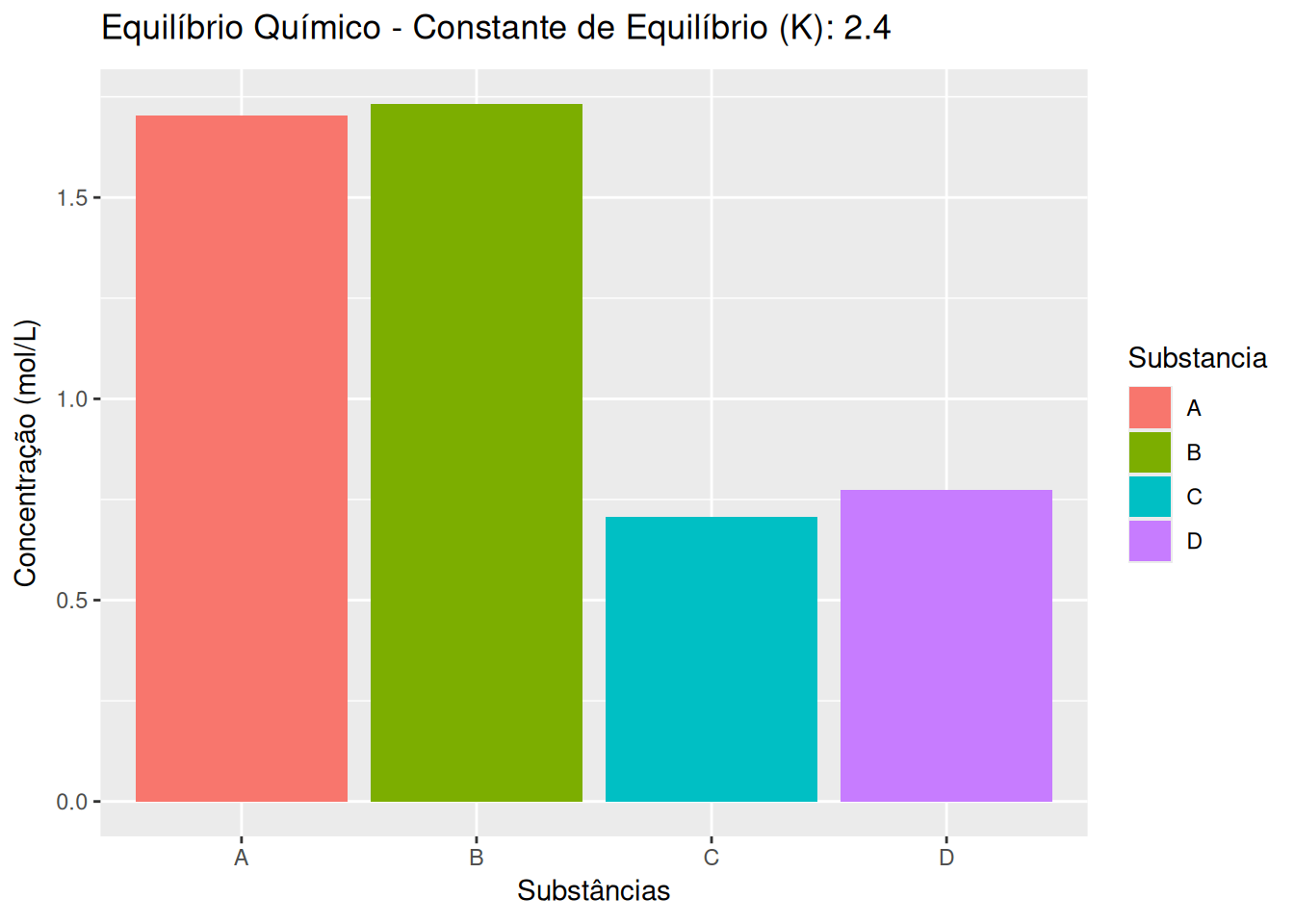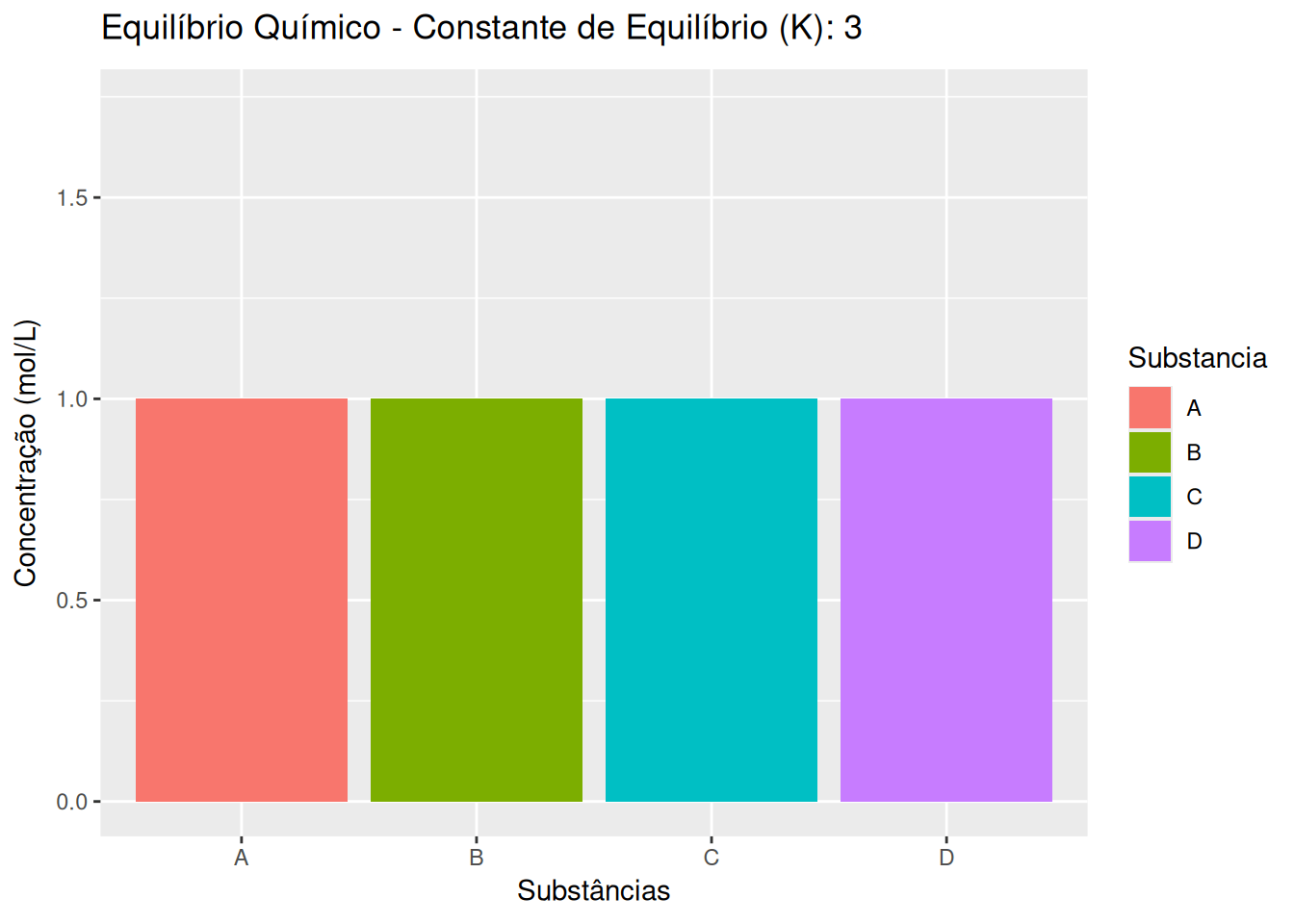
7 Concentração de reagentes e constante de equilíbrio de dissociação (EM13CNT201, EM13CNT202, EM13CNT204)
# Carregar pacotes necessários
library(ggplot2)
library(gganimate)
# Definir os parâmetros iniciais
A <- 1 # Concentração de A
B <- 1 # Concentração de B
# Função para calcular as concentrações de produtos com base na constante K
calc_produtos <- function(K) sqrt(K * A * B)
# Criar o conjunto de dados para diferentes valores de K
dados <- data.frame(
K = seq(0.5, 3, by = 0.1)
)
dados$C <- calc_produtos(dados$K)
dados$D <- dados$C
# Transformar dados para visualização de barras
dados_long <- data.frame(
K = rep(dados$K, each = 4),
Substancia = rep(c("A", "B", "C", "D"), times = length(dados$K)),
Concentracao = c(rep(A, length(dados$K)), rep(B, length(dados$K)), dados$C, dados$D)
)
# Criar gráfico animado
p <- ggplot(dados_long, aes(x = Substancia, y = Concentracao, fill = Substancia)) +
geom_bar(stat = "identity", position = "dodge") +
labs(title = 'Equilíbrio Químico - Constante de Equilíbrio (K): {closest_state}',
x = 'Substâncias', y = 'Concentração (mol/L)') +
transition_states(K, transition_length = 2, state_length = 1) +
ease_aes('linear')
# Mostrar o gráfico
pSugestões:
Experimente modificar o gráfico, usando/substituindo alternativamente os comandos abaixo no trecho de código:
scale_fill_manual(values = c("A" = "blue", "B" = "green", "C" = "red", "D" = "purple")) # Definida uma paleta de cores personalizada